Wielokrotnie powtórzone sekwencje centromeropodobne parS w replikonach z rodziny repABC
17 05 2022
Kategoria: Seminarium IM
Zapraszamy na seminarium instytutowe: w najbliższy poniedziałek 23 maja o godz. 14 mgr Elvira Chapkauskaitse z Zakładu Genetyki Bakterii wygłosi seminarium pt. „Wielokrotnie powtórzone sekwencje centromeropodobne parS w replikonach z rodziny repABC: plazmid pAMI4 Paracoccus aminophilus JCM 7686 jako model badawczy”.
Streszczenie:
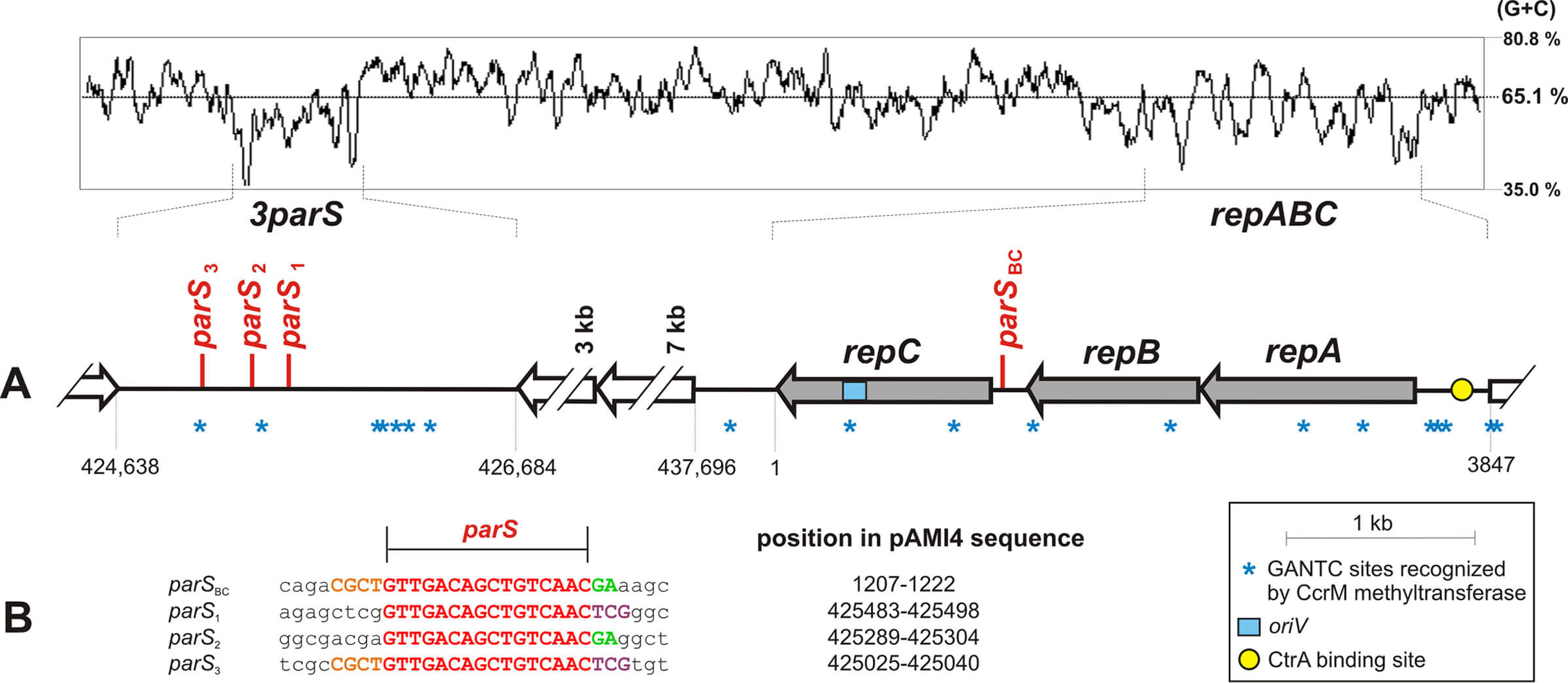
Systemy partycyjne (par) umożliwiają stabilne utrzymywanie pozachromosomowych replikonów, które występują w komórce bakteryjnej w niskiej liczbie kopii. Loci te składają się z dwóch genów kodujących białka partycyjne A i B oraz z sekwencji centromeropodobnej parS. Systemy partycyjne są dosyć często zlokalizowane w bliskim sąsiedztwie z systemami replikacyjnymi. Ciekawym przykładem takiego współwystępowania są replikony repABC z klasy Alphaproteobacteria, w których geny partycyjne (repAB) oraz replikacyjne (repC) tworzą jeden operon, z sekwencją centromeropodobną parS położoną w pobliżu tych genów. Analizy genomiczne plazmidu pAMI4 (438 kpz), pochodzącego z Paracoccus aminophilus JCM 7686, wykazały, że replikon ten oprócz operonu repABC (z parS znajdującą się między genami repB i repC) posiada obszar niekodujący o wielkości ok. 2 kpz, zawierający trzy dodatkowe powtórzenia parS. Głównym celem niniejszych badań było sprawdzenie, czy locus 3parS jest zaangażowane w stabilne utrzymywanie się plazmidu pAMI4 (tj. maintenance).
Udowodniliśmy, że locus 3parS wiąże się z białkiem partycyjnym RepB w warunkach in vitro oraz pokazaliśmy, że region ten jest niezbędny do odpowiedniej segregacji kopii plazmidu pAMI4 do komórek potomnych in vivo. W celu identyfikacji podobnych loci, przeprowadzono analizy porównawcze dystrybucji sekwencji centromeropodobnych w innych replikonach repABC. Wykazały one odmienne wzorce występowania dodatkowych powtórzeń sekwencji parS w plazmidach i chromidach bakterii z rzędów Rhodobacterales i Rhizobiales. Wiele z tych powtórzeń parS były zlokalizowane w obrębie otwartych ramek odczytu, np. wewnątrz genów cas systemu CRISPR-Cas.
Podsumowując, zidentyfikowaliśmy nowe locus, które jest wymagane do właściwej partycji plazmidu repABC. Wyniki tych badań stawiają pytania dotyczące biologicznych przyczyn zróżnicowanej dystrybucji sekwencji parS, która może skutkować zmianami w regulacji ekspresji operonu repABC, jak również innymi trybami procesów replikacji i partycji replikonów z rodziny repABC.
