Rozwój wysokoprzepustowej metody scRNA-seq dla bakterii
31 05 2023
Kategoria: Seminarium IM
Zapraszamy na seminarium instytutowe: w poniedziałek 5 czerwca o godz. 14 w sali 102B mgr Marcin Małkowski z Zakładu Mikrobiologii i Biotechnologii Środowiskowej wygłosi seminarium pt. „Opracowanie nowatorskiej, wysokoprzepustowej metody sekwencjonowania pojedynczych komórek bakterii do badania społeczności drobnoustrojów i interakcji gospodarz-patogen”.
Streszczenie:
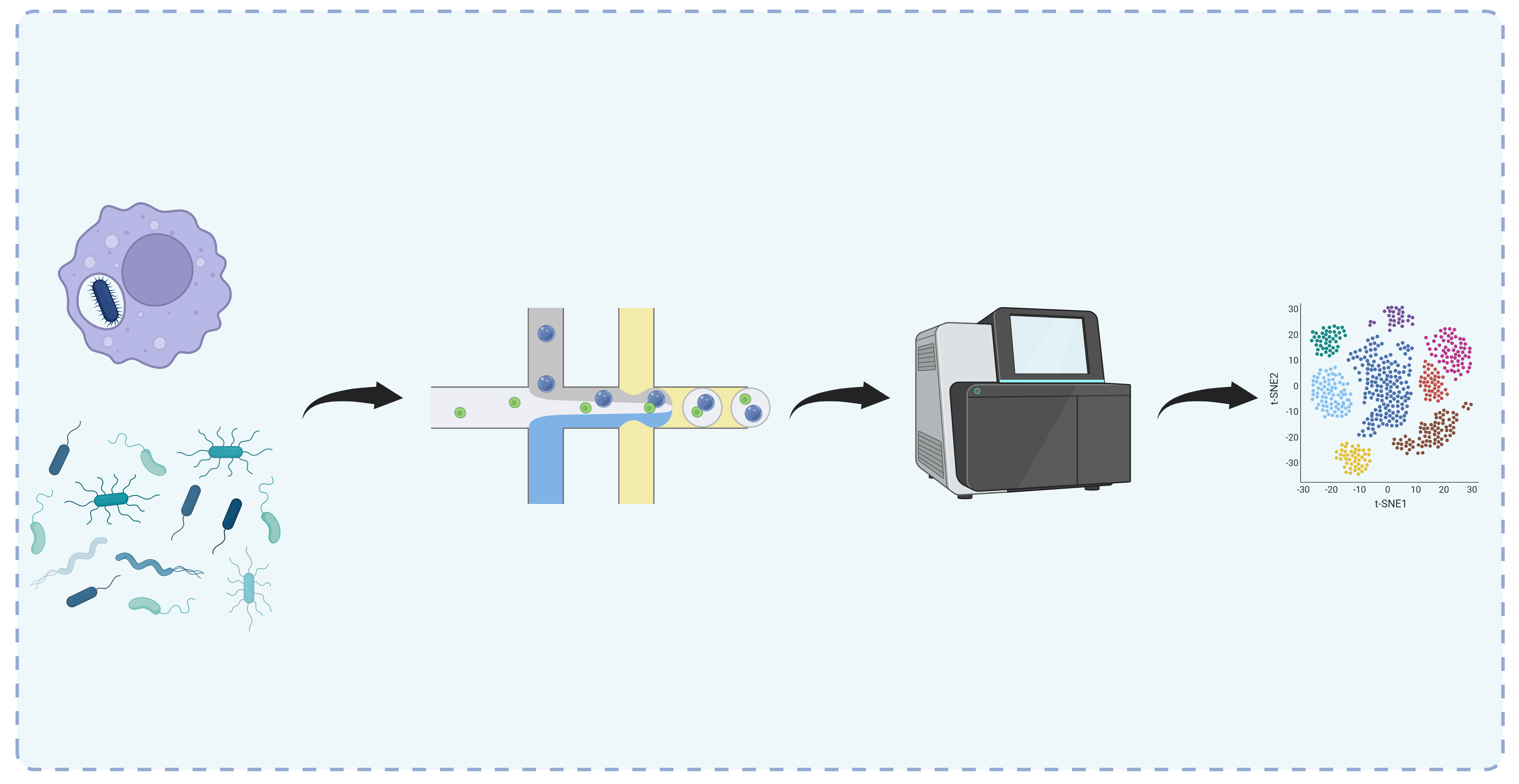
Rozwój technologii sekwencjonowania pojedynczych komórek obecnie rewolucjonizuje biologię i medycynę, pozwalając przede wszystkim bardziej szczegółowo badać transkryptom komórek. Odkrywane są nowe typy i stany komórek, tworzone są atlasy komórkowe, a liczba komórek jaką można analizować ciągle rośnie. W przypadku badań komórek eukariotycznych istnieje wiele metod, które pozwalają efektywnie zbadać ich transkryptom, jednak ze względu na znaczące różnice w budowie i cech RNA komórek prokariotycznych, metody te trudno jest stosować do komórek bakterii. Celem tego projektu jest rozwój, optymalizacja oraz aplikacja wysokoprzepustowej metody profilowania całego transkryptomu VASA-seq, oraz jej wdrożenie do analiz pojedynczych komórek bakteryjnych. Kolejnym etapem projektu będzie przetestowanie uniwersalnego protokołu do analiz zarówno bakterii Gram-dodatnich i Gram-ujemnych, dzięki czemu w dalszej perspektywie metoda ta będzie pozwalała na badania mikrobiologicznych konsorcjów, a nawet komórek eukariotycznych i bakterii jednocześnie np. makrofagów zainfekowanych patogenami wewnątrzkomórkowymi.
