Nowe narzędzia genomiczne
25 05 2022
Kategoria: Seminarium IM
W poniedziałek 30 maja o godz. 14.00 mgr Mikołaj Dziurzyński z Zakładu Mikrobiologii i Biotechnologii Środowiskowej przedstawi seminarium pt. „Opracowanie nowych narzędzi do genomicznych i meta genomicznych analiz bakterii metaloopornych”. Serdecznie zapraszamy!
Streszczenie:
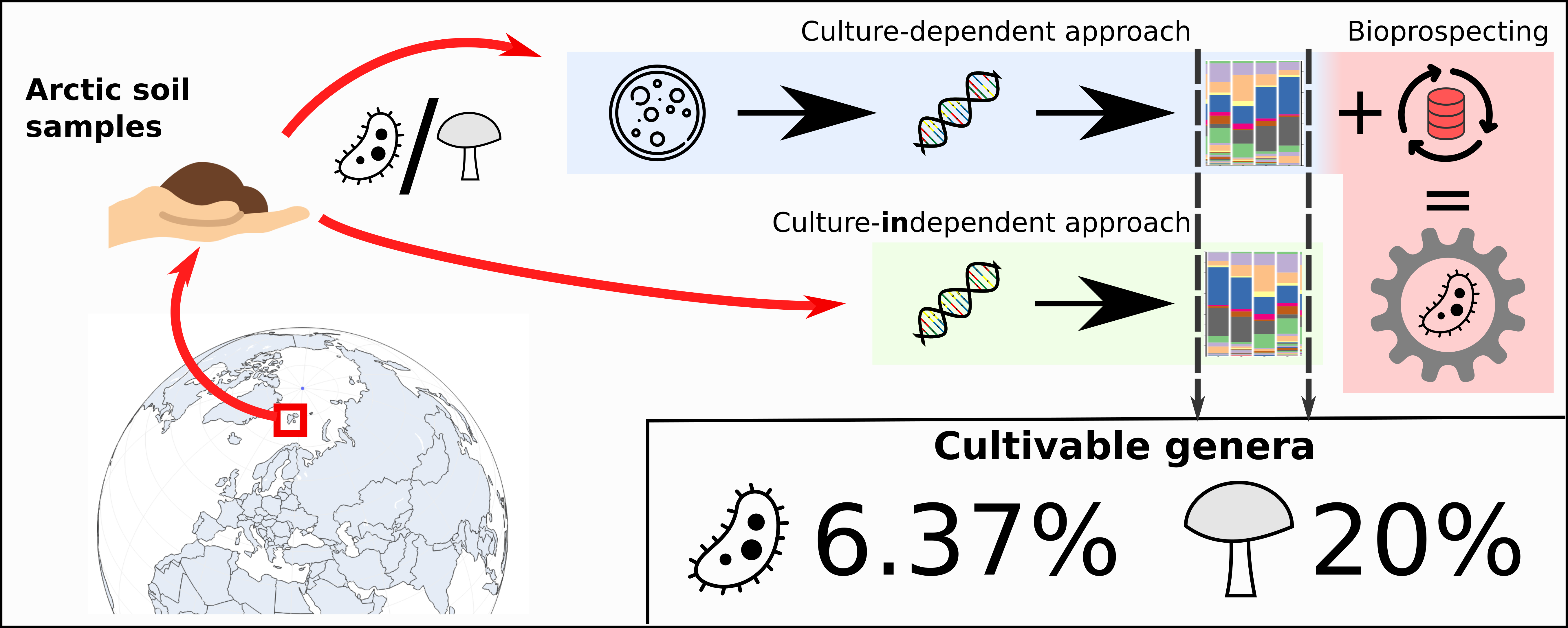
W ramach niniejszej pracy doktorskiej opracowano nowe narzędzia bioinformatyczne, przeznaczone do analizy genów związanych z metabolizmem i opornością bakterii na metale. Mikrobiomy arktyczne, ze względu na ich unikatowość i wysoki potencjał biotechnologiczny zostały wykorzystane do opracowywania, testowania i walidacji tych narzędzi.
Badania umożliwiły szczegółowy wgląd w różnorodność gleb arktycznych i po raz pierwszy oszacowały jaką część mikrobioty występującej w tym środowisku można hodować z użyciem standardowych metod laboratoryjnych. Wyniki wykazały, że standardowe metody hodowli znacząco zaburzają obserwowaną różnorodność badanych próbek i są w stanie odzyskać tylko niewielką część rodzajów bakteryjnych i grzybowych zaobserwowanych przy użyciu metod niehodowlanych. Wykazano, że w bioprospektywnych analizach gleb arktycznych należy stosować specjalne, ukierunkowane protokoły hodowlane. W ramach tej pracy zaproponowano i przetestowano jeden taki protokół. Protokół opierał się na stosunkowo niedrogiej analizie różnorodności mikrobiologicznej i wykorzystaniu danych genomowych z publicznych repozytoriów. Nowy protokół został opracowany w celu zwiększenia szansy izolacji bakterii wielometaloopornych na potrzeby przyszłych technologii biormediacyjnych.
W ramach pracy opracowano również nowe narzędzia bioinformatyczne. W celu zwiększenia wydajności manualnej adnotacji bakteryjnego DNA opracowano aplikację MAISEN. MAISEN umożliwia użytkownikom adnotację interesujących ich sekwencji za pomocą intuicyjnego interfejsu i jest dostępny nieodpłatnie, za pośrednictwem przeglądarki internetowej. MAISEN pozwala na adnotację nowo zsekwencjonowanych genomów, a także na readnotację sekwencji już opisanych. Głównym celem opracowania systemu MAISEN było skrócenie czasu potrzebnego na dokładną manualną adnotację sekwencji DNA poprzez zapewnienie użytkownikom wszechstronnego narzędzia dostarczającego wyniki przyrównań sekwencji z różnych baz danych. Dzięki takiemu podejściu użytkownik może skupić się na adnotacji swojej sekwencji, zamiast poświęcać czas na obsługę wielu różnych narzędzi i baz danych.
Stworzono również bazę danych LCPDb-MET, zawierającą rankingi par starterów do reakcji PCR, zaprojektowanych do wykrywania genów związanych z metabolizmem i opornością bakterii na metale. Analiza wykazała, że przytłaczająca liczba par starterów dostępnych w literaturze naukowej nie powinna być wykorzystywana do wykrywania docelowych genów w badaniach środowiskowych. Ponieważ ocena jakości par starterów przeprowadzona w ramach niniejszej pracy wymagała dokładnej bazy referencyjnej, stworzono nową bazę danych (MetGeneDb), proponującą dodatkowy poziom zróżnicowania dla sekwencji genów związanych z metabolizmem i opornością bakterii na metale.
